
致病性大肠杆菌和志贺氏菌是发展中国家儿童腹泻死亡和全球食源性感染的主要原因,其抗生素耐药性日益严峻,对公共卫生构成重大威胁。
而AI的突破性进展,为这一棘手问题带来新的曙光。
近日,来自莫纳什大学、墨尔本大学等机构的团队在Nature Communications上发布了题为Inhibiting heme piracy by pathogenic Escherichia coli using de novo-designed proteins的论文。

科学家们利用Alphafold、RFdiffusion、ProteinMPNN这些来自诺奖团队的顶级模型,从头设计出可立即使用的新型蛋白质,能够杀死大肠杆菌等抗生素耐药细菌,为全球日益严峻的超级细菌威胁提供了创新解决方案。
这项研究不仅为开发针对耐药菌株的药物提供了全新机制,更凸显了人工智能在整合膜蛋白抑制剂设计中的巨大潜力,预示着感染性疾病治疗领域的新突破。
这项成果基于莫纳什生物医学发现研究所和墨尔本大学Bio21研究所共同开发的AI蛋白质设计项目(AIPDP),该项目由经过验证的公开软件(AlphaFold、RFDiffusion等)和定制网络组成,并包含了一支由结构生物学家和计算机科学家组成的专家团队。
目前AIPDP已经开发了一套端到端的蛋白质工程平台,能够构建具有多样化的蛋白质分子,用于开发药物、疫苗、纳米材料及微型传感器,展示出广阔的应用前景。
AI“从零”设计,告别低效试错
铁是几乎所有细菌必需的营养元素,参与DNA合成、能量代谢等关键生理过程。在感染过程中,宿主通过多种机制严格限制游离铁的可利用性,这一防御策略被称为"营养免疫"。
为突破这一限制,病原菌进化出两种途径:分泌铁载体捕获铁或直接摄取宿主的血红素。这两种方式都依赖外膜蛋白TonB依赖性转运体(TBDT)。
其中,血红素转运蛋白ChuA能快速从血红蛋白中提取血红素,其关键组氨酸残基(His-86和His-420)对这一过程至关重要,但具体机制尚不清楚。
研究团队利用结构建模、冷冻电镜、X射线晶体学、诱变和表型分析的组合来确定该过程的机制细节。
其中包括利用AlphaFold2-Multimer预测了ChuA与血红蛋白的相互作用模式,并通过AlphaFold3对ChuA-血红素复合物的建模预测了His-420和His-86共同配位血红素的可能性,为理解这两个残基的协同作用提供了结构基础。
凭借这些结构信息,团队采用RFdiffusion生成了5000个潜在的结合蛋白骨架构象。这些初始设计随后进入深度学习优化流程,ProteinMPNN为每个骨架设计了最优氨基酸序列,AlphaFold2则通过pAE相互作用分数评估设计质量。
最终,基于结构多样性、结合界面特性和实验可行性等标准,研究人员精选出96个最优设计进入实验验证阶段,其中最具潜力的四个蛋白(A10、C8、G7和H3)表现出显著的抑制效果。
特别值得注意的是,这些抑制蛋白具有高度特异性,只影响血红蛋白依赖的生长途径,而对其他铁获取机制没有干扰。
结构生物学研究进一步验证了AI设计的准确性,冷冻电镜清晰展示了G7和H3与ChuA形成的复合物结构,与AlphaFold2模型预测结果高度吻合。
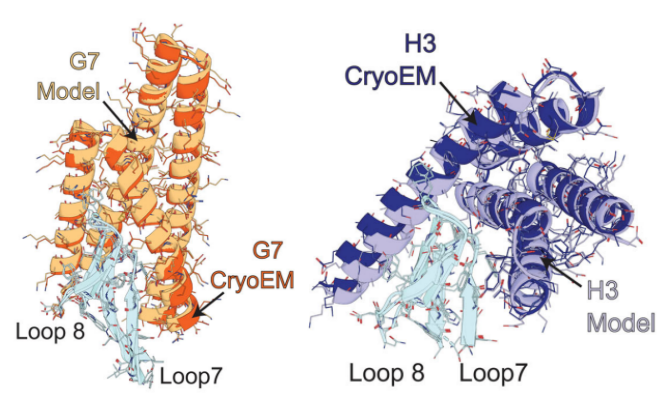
这项研究最引人注目的发现在于,部分AI设计的蛋白质(如G7)无需任何实验优化就直接表现出纳摩尔级的抑制活性。这种“一次设计成功”的现象打破了传统蛋白质设计中需要多轮试错的瓶颈。
该团队证实了AI设计的蛋白质能有效阻断致病性大肠杆菌通过ChuA获取铁元素的途径。更重要的是,他们建立了一个可推广的AI蛋白质设计方法。
这些发现为AI在生物医学领域的应用开辟了新的可能性,突破了传统药物设计的局限,为治疗耐药菌感染等问题提供了新思路。
集成尖端AI工具,赋能蛋白质设计
上述成果成为AI蛋白质设计项目(AIPDP)的又一力作,此前该团队已经利用RFDiffusion和ProteinMPNN设计了可溶性细菌酶和细菌膜蛋白的抑制剂。
AIPDP致力于整合AI驱动的蛋白质设计工具,帮助研究人员快速掌握并应用这些技术,推动澳大利亚在该领域的研究与创新。
据悉,这是该国首个引入David Baker实验室诺奖级蛋白质设计技术的创新平台,其开发了一种端到端蛋白质工程方法,可用于开发药物、疫苗、纳米材料和微型传感器。
AIPDP拥有一支专业的结构生物学家和计算机科学家团队,由Rhys Grinter博士和Gavin Knott副教授共同领导。

图:Rhys Grinter(左)和Gavin Knott(右)
Rhys Grinter博士毕业于格拉斯哥大学,在应用结构生物学、分子细胞生物学和化学生物学综合方法方面拥有丰富经验,现任墨尔本大学Bio21分子科学与生物技术研究所Grinter实验室的负责人。
Gavin Knott为莫纳什生物医学发现研究所副教授,其研究聚焦RNA结合蛋白的进化机制及CRISPR系统,其负责的Knott实验室专注于计算生物学、生物信息学、生物化学、结构生物学和细胞生物学的交叉创新。
目前,AIPDP正在打造一个越来越大的技术平台和生态,团队正在不断测试、评估和比较新的工具和软件(如Bindcraft和Chai等),根据具体的蛋白质设计问题定制使用方案,为学术界和产业界提供AI蛋白质设计服务,加速开发新型疗法、诊断工具和科研应用。
Grinter博士表示,深度学习新方法能够高效地从头设计具有特定特性和功能的蛋白质,从而降低成本并加速新型蛋白质结合剂和工程酶的开发。
推荐阅读